 |
Über die Entstehung neuer Proteine
von Matti Leisola
Studium Integrale Journal
11. Jahrgang / Heft 2 - Oktober 2004
Seite 67 - 75
|
 |
Zusammenfassung: Proteine sind unverzichtbare Bestandteile aller bekannten Lebewesen. Sie erfüllen unterschiedlichste Aufgaben. Sie stellen wichtige strukturelle Moleküle, Baustoffe von Zellen dar oder wirken als molekulare Maschinen oder Biokatalysatoren. Durch die Erkenntnisse der Molekularbiologie sind manche Zusammenhänge zwischen Struktur und zugehöriger Funktion aufgedeckt worden.
In diesem Beitrag werden Vorstellungen, wie die unterschiedlichsten Proteinstrukturen und damit auch deren Funktionen durch zufällige Prozesse entstanden sein könnten, kritisch geprüft und mit empirischen Befunden aus der Protein-Biochemie verglichen. Dabei wird erkennbar, daß bisher keine überzeugenden Mechanismen für eine zufällige Entstehung von funktionellen Proteinen beschrieben sind und daß selbst die Entstehung neuer Funktionen auf der Basis bereits bekannter Strukturen nur in sehr eingeschränktem Maße nachvollziehbar sind bzw. in der Biotechnologie genutzt werden können.
|  |
Proteine als funktionelle Moleküle
Proteine weisen ein breites Spektrum an biologischen Funktionen auf. Das grundlegende Kennzeichen von Proteinen ist ihre Funktionalität. Man könnte sie als „intelligente Moleküle“ bezeichnen. Unsere Haare bestehen aus einem bestimmten Proteintyp. Unsere Knochen, Nägel, Sehnen und Muskeln sind aus Proteinen aufgebaut. Diese werden als Strukturproteine bezeichnet. Sie sind auch in den Zellmembranen, Zellwänden und inneren Strukturen von Zellen von Bedeutung.
Neben strukturellen Funktionen erfüllen Proteine in Organismen noch viele andere Aufgaben. Viele Hormone sind Proteine, ebenso Toxine. Unser Immunsystem nutzt Proteine, die als Antikörper bezeichnet werden, um körperfremde Moleküle, Viren und sogar Bakterien anzugreifen. Proteine sind an der Kommunikation zwischen Zellen beteiligt und sie stellen Schlüsselkomponenten in Viren dar. Proteine sind auch verantwortlich für Bewegungsabläufe bei Organismen. Kleine Elektromotoren in Bakterien sind aus Proteinen aufgebaut. Muskelaktivität basiert auf der Fähigkeit von Proteinfilamenten, aneinander zu gleiten. Resilin, ein sehr elastisches Protein, ermöglicht Flöhen ihre Sprünge. Spinnenseide besteht aus Proteinen, die elastisch sind und stärker als Stahl.
Proteine dienen außerdem als Katalysatoren. Biologie wäre ohne sie gar nicht möglich. Alle biologischen Aktivitäten benötigen Katalysatoren. Diese Aktivitäten werden zusammenfassend als Stoffwechsel bezeichnet. Katalytisch aktive Proteine sind Enzyme. Stoffwechselreaktionen sind wichtig, um die Nahrungsmittel, die wir essen, abzubauen. Enzyme katalysieren die Zellteilung und die Synthese von DNA und sogar die von Proteinen.
 |
Abb. 1: Vom Gen zum Protein. Die Sequenz der DNS-Basen (A) wird von einem komplizierten Enzymkomplex, der RNS-Polymerase, kopiert (B): es entsteht messenger-RNS (mRNS). Ribosomen lagern aminosäuretragende transfer-RNS-Moleküle (tRNS) an die Basentripletts der mRNS an (C). Die Aminosäuren werden dort zu einer wachsenden Kette verbunden (D). Die entstehende Aminosäurekette faltet sich dann zum funktionalen Protein zusammen (E). |
|
|
|
Struktur und Biosynthese von Proteinen
Die Biosynthese von Proteinen erfolgt durch einen raffinierten Prozeß. Proteine werden aus etwa zwanzig Bausteinen aufgebaut – den Aminosäuren. Die Aminosäuren haben unterschiedliche Eigenschaften. Einige sind sauer, andere neutral oder basisch, wieder andere sind aromatisch. Proteine sind aus Aminosäuren aufgebaut ähnlich wie Wörter aus Buchstaben aufgebaut sind. Die Abfolge (Sequenz) bestimmt die Funktion. Die Information zur Proteinherstellung liegt in der DNA. Von dieser Information wird eine Kopie, die mRNA (messenger RNA), erstellt, die ihrerseits als Anleitung zur Proteinsynthese in Ribosomen verwendet wird (Abb. 1). Ein durchschnittliches Protein besteht aus etwa 300 Bausteinen, die miteinander durch sogenannte Peptidbindungen chemisch verknüpft sind. Ein typisches Protein besteht nicht einfach aus einer langen, linearen Kette von Aminosäuren. Proteine haben eine dreidimensionale Struktur, man unterscheidet Primär-, Sekundär-, Tertiär- und Quartärstrukturen (Abb. 2).
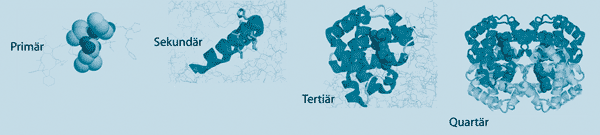 |
| Abb. 2: Beispiele für Primär-, Sekundär-, Tertiär- und Quartärstrukturen von Proteinen (v. l. n. r.). |
|
Manchmal falten sich Proteine spontan in diese 3-D-Struktur, aber in vielen Fällen unterstützen Helferproteine (Chaperone) die Faltung in die richtige Struktur. Diese korrekte Struktur eines Proteins ist unbedingte Voraussetzung für seine Funktion. Falsch gefaltete Strukturen haben gewöhnlich den Verlust der Funktion zur Folge. Die Veränderung von Eiweiß beim Kochen oder bei der Käseherstellung sind alltägliche Beispiele für Inaktivierung und Zusammenlagerung von Proteinen.
Proteine sind hauptsächlich aus zwei unterschiedlichen Sekundärstrukturelementen aufgebaut, die so genannten b-Falblätter und a-Helices. Die Form eines Proteins wird als Faltung bezeichnet. Schätzungen über die die Gesamtzahl möglicher Faltungen variieren zwischen 1.000 und 10.000 (Leonov et al. 2003, Rost 2002). Drei Beispiele solcher Proteinfaltungen sind in Abb. 3 dargestellt.
|
Das Konzept des Sequenzraums wurde zuerst von Rechenberg (1973) eingeführt und später von Swetina & Schuster (1982), Feistl & Ebeling (1982) und Eigen (1985) angewendet (Eigen entwickelte in diesem Konzept das „Quasi-Spezies“-Modell). Im Sequenzraum werden Genotypen (z.B. Nukleinsäure- oder Proteinsequenzen) als Punkte in einem n-dimensionalen Raum repräsentiert. Das theoretische Konzept des Sequenzraums erlaubt eine mathematische – nicht mehr unmittelbar anschauliche – Behandlung der ungeheuer großen Zahl aller möglichen Sequenzen. Für eine Nukleinsäuresequenz aus 100 Nukleotiden sind es 4100 (1,6 x 1060) Raumpunkte. Mathematisch kann man zeigen, daß bei zunehmender Zahl der Raumpunkte der Abstand zwischen den einzelnen Punkten reduziert wird (Eigen 1985). Auf diesen Zusammenhang gründet sich die Idee, daß unter der unübersehbar großen Zahl möglicher Sequenzen ein Weg von einer funktionellen Sequenz zu einer anderen so klein wird, daß die Wahrscheinlichkeit, daß er durch Zufallsprozesse realisiert wird, in vorstellbare Größenordnungen rückt.
Im Text wird gezeigt, daß bisher die empirischen Befunde diese Überlegung nicht unterstützen.
|
|
|
Sequenzraum von Proteinen
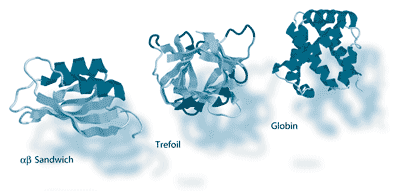 |
| Abb. 3: Drei unterschiedliche Faltungstypen bei Proteinen. Die Struktur von Proteinen wird üblicherweise durch Beugung von Röntgenstrahlen an Proteinkristallen aufgeklärt. |
|
Proteine können sehr unterschiedlich aufgebaut sein. Dieser riesige Bereich von Möglichkeiten wird Sequenzraum genannt. Ein Protein aus 100 Aminosäuren kann in 20100 (1,3 x 10130) verschiedenen Varianten aufgebaut werden. Das ist eine schwindelerregende Zahl. Wir können diese Angabe mit der Masse der Erde vergleichen, die etwa 6 x 1027 g beträgt. Wenn diese Masse aus Proteinen von je 100 Aminosäuren bestünde, ergäbe das ungefähr 3 x 1047 verschiedene Sequenzen. Diese große Zahl unterschiedlicher Proteine würde aber nur einen kleinen Ausschnitt aller möglichen Anordnungen darstellen.
Stellen wir uns die Herstellung von Proteinen durch zufällige Prozesse vor, dann müssen wir die Frage stellen: Wie häufig sind funktionale Proteine im Sequenzraum? In einem kürzlich erschienenen Artikel ist dieses Problem anschaulich dargestellt (Abb. 4; Bolon et al. 2002).
|
|
Theorien zur Evolution von Proteinen
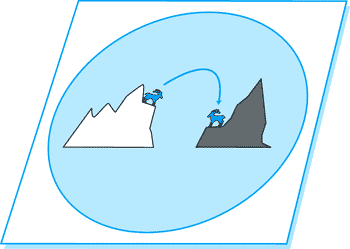 |
| Abb. 4: Um ein Protein mit der Funktion A in eines mit der Funktion B zu verändern, muß eine große Distanz in einem unbekannten Sequenzraum übersprungen werden, wenn die neue Funktion nicht bereits im ursprünglichen Protein vorhanden war. |
|
Nach evolutionären Vorstellungen ist es selbstverständlich, daß verschiedene Organismen von einem gemeinsamen Vorfahren abstammen. Mehrere Beweislinien werden angeführt, um diese Annahme zu unterstützen. Eine davon ist die Ähnlichkeit der grundlegenden Biochemie in allen Lebewesen. Die Systeme zum Speichern und Abrufen der Erbinformation und die Maschinerie zur Proteinherstellung sind in Grundzügen in allen Organismen dieselben. Deshalb kann man die Erbinformation für Humaninsulin (ein Protein aus 51 Aminosäuren) benützen, um Insulin in Bakterien oder Hefe herzustellen. Nach evolutionstheoretischen Vorstellungen lassen sich alle Proteine von einem Vorläuferprotein mit ähnlicher Struktur und Funktion ableiten:
„Jedes Protein wurde durch schrittweise Verbesserung und Verfeinerung der Funktion über Millionen von Jahren angepaßt ... Weil Anpassung so lange dauert, ist es unwahrscheinlich, daß brauchbare Proteine fertig aus zufälligen DNA-Sequenzen oder aus Pseudogenen entstehen. Stattdessen entwickeln sie sich durch Anlagerung, Aufsetzen von neuen Funktionen auf alte, und neue strukturelle Merkmale, die in neuen Zusammenhängen lernen“ (McLachlan 1987).
 |
| Unterschiedliche Proteine ergeben verschiedene Ähnlichkeitsbäume, die häufig im Widerspruch zu traditionellen Stammbäumen stehen. |
 |
|
Die Gruppe der Globine ist ein typisches Beispiel für schrittweise Proteinevolution. Man nimmt an, daß alle sauerstoffbindenden und -transportierenden Hämoglobine und Myoglobine sich von einer gemeinsamen Grundstruktur ableiten. Evolution soll durch zufällige genetische Veränderungen und natürliche Auslese vorangetrieben worden sein. Vor diesem Hintergrund vertritt man die Annahme, daß sich Proteine ähnlicher Funktion im Laufe der Zeit von einem Lebewesen zum nächsten verändern. Auf der Grundlage der Unterschiede von Proteinen oder anderen informationstragenden Molekülen von Zellen haben Wissenschaftler evolutionäre (phylogenetische) Stammbäume entworfen. Solche Bäume werden aufgrund der großen Datenmenge von Computerprogrammen nach verschiedenen Algorithmen berechnet. Vor zwanzig Jahren dachte man, daß solche molekularen Ähnlichkeitsbäume die Evolution ohne jeden Zweifel beweisen. Inzwischen wissen wir, daß unterschiedliche Proteine verschiedene Ähnlichkeitsbäume ergeben, die häufig im Widerspruch zu traditionellen Stammbäumen stehen, welche aufgrund der Morphologie oder auf der Basis des Fossilbefundes erstellt werden (Morris 2000).
Proteine ändern sich, wenn sich ihre genetische Vorlage verändert. Es sind viele verschiedene Mechanismen oder Arten von Veränderung bekannt, die Änderungen in der Struktur der Proteine verursachen (Tab. 1).
|
|
Theorie der Genduplikation
Man geht allgemein davon aus, daß heutige Gene durch Genduplikation mit nachfolgender Veränderung aus einem kleinen Bestand von sehr alten Genen hervorgegangen sind (Abb. 5) (Ohno 1970). Neue funktionelle Proteine sollen sich durch Mischen (shuffling) von grundlegenden Proteinbereichen zu neuen Kombinationen entwickelt haben. Indirekte Belege für diese Hypothese beruhen auf der Tatsache, daß viele verschiedene Proteine unterschiedlicher Funktion aus ähnlichen Bauelementen aufgebaut sind (a/b-Faß in Xylanase, Xylose-Isomerase, Triosephosphat-Isomerase, Cyclodextrin-Glycosyltransferase etc.). Außerdem findet man in der Natur häufig „Mosaik-Proteine“, das sind Proteine, die aus vielen (oft sich wiederholenden) Domänen aufgebaut sind.
 |
| Tab. 1: Genetische Veränderungen, die zu veränderten Proteinen führen. |
|
Durch Verknüpfen der Theorie der Genduplikation mit der Erkenntnis, daß eine Vielzahl von Sequenzen ähnliche Strukturen annehmen können, vermeidet man die sehr geringe Wahrscheinlichkeit der Entstehung neuer (ähnlicher) Gene aus Zufallssequenzen (Lau & Dill 1990, Golding et al. 1994). Nach dieser Sichtweise sollen sich also neue Gene durch Modifikation von bereits existierenden entwickeln.
Oberflächlich betrachtet scheint die Theorie der Genduplikation das Problem der zufälligen Suche nach funktionsfähigen Sequenzen im Sequenzraum zu lösen. Diese Theorie sieht sich aber mit verschiedenen Problemen konfrontiert.
- Erstens bringt Selektion nicht effektiv neue Proteine hervor, tendenziell bewahrt sie vorhandene Funktionen oder verbessert diese. Allgemein ist die Tendenz zu erkennen, daß Organismen auf einem lokalen Optimum in einer bergigen Fitness-Landschaft stecken bleiben, was zu evolutionärer Stasis (Stillstand) führt (McFadden & Knowles 1997).
- Zweitens zeigen Computersimulationen, daß der Austausch einzelner Aminosäuren nicht effektiv ist für die Bildung neuer Proteine (Stemmer 1994, Bogarad & Deem 1999). Daher müßten größere Blöcke von Proteinsequenzen neu angeordnet werden, um Proteine mit neuen Strukturen zu erzeugen. Im Labor können Proteine in Stücke gespalten und neu kombiniert werden (DNA-shuffling), in der Natur ist ein solcher Vorgang aber selten; wenn er sich ereignet, sind funktionale Proteine sehr selten zu erwarten. Eine solche Neubildung von Bausteinen scheint heutzutage selten zu sein (Ganfornina & Sánchez 1999).
- Drittens verfügen tetraploide Frösche über ein doppeltes Genom, aber die duplizierten Gene haben sich während der vermuteten Jahrmillionen dauernden Evolution kaum verändert (Hughes & Hughes 1993).
- Viertens ist von vielen Arten bekannt, daß sie ihr Genom von zusätzlicher DNA reinigen (Petrov et al. 1996, Mira et al. 2001).
- Fünftens können an einem Protein 50% seiner Aminosäuren oder mehr ausgetauscht werden, ohne daß die ursprüngliche Aktivität geändert wird.
- Sechstens werden bei einer schrittweisen Evolution von neuen Proteinen sehr wahrscheinlich auch Phasen durchlaufen, in denen das Protein keine Funktion besitzt und damit einem geringen Selektionsdruck unterliegt; dies führt gewöhnlich zu einer Anhäufung von inaktivierenden Mutationen. Um über eine Distanz von nicht vorhandener Funktionalität mit einem riesigen evolutionären Sprung zu springen, benötigt man so viele Änderungen in einem einzigen Schritt (oder in wenigen Schritten), daß die Herausforderung ähnlich groß ist, wie eine neue Funktion zufällig im Sequenzraum zu finden.
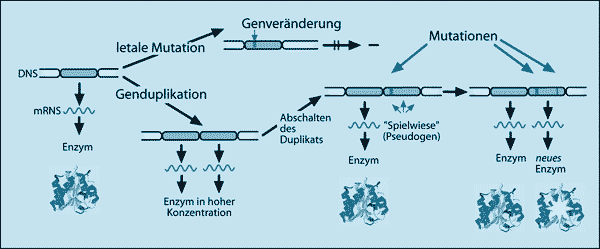 |
| Abb. 5: Die Theorie der Genduplikation geht davon aus, daß Kopien von bereits vorhandenen Genen frei mutieren können (das trifft aber nicht immer zu) und neue Enzyme bilden können (Annahme). |
|
Diese Überlegungen führen zum Schluß, daß evolutionäre Mechanismen zwar in der Lage sind, eine bestehende Funktion zu verbessern, daß es aber sehr unwahrscheinlich ist, daß ein evolutionärer Prozeß neue Proteine mit einer Geschwindigkeit erzeugen kann, die erforderlich wäre, um Evolution größeren Ausmaßes zu ermöglichen. Der Sequenzraum scheint hinsichtlich funktioneller Proteine eigentlich leer zu sein, und ein zufälliger Gang von einer Proteinfaltung oder Enzymaktivität zur andern scheint sowohl theoretisch als auch experimentell sehr schwierig zu sein, sogar wenn man von einem funktionellen Protein ausgeht.
Es ist bekannt, daß funktionelle Veränderungen, die durch Austausch an wenigen Aminosäurepositionen verursacht werden, in der Natur häufig vorkommen. Diese Art von Mikroevolution läuft innerhalb des Sequenzraums ab, der an modernen Organismen effektiv getestet werden kann. Bei natürlichen Vorgängen kann der Austausch von Aminosäuren sogar neue Aktivitäten im Rahmen der bereits bestehenden Proteinfaltung und -funktion hervorrufen. Wenn jedoch der Sequenzraum einer neuen Proteinfaltung die Grenzen der ursprünglichen Faltung überschreitet, sind dem Proteinrepertoir, das in natürlichen Organismen mit Hilfe evolutionärer Prozesse erzeugt werden kann, enge Grenzen gesetzt (Evolution im Reagenzglas).
Neue Proteinfaltungen. Aminosäureaustausche können die Entstehung neuer Proteinfaltungen nicht ausreichend bewirken und erklären. Deshalb wurde vorgeschlagen, daß Rekombination von Proteinteilen in der Erzeugung von Proteinen mit neuen Strukturen und Funktionen wirksamer ist und daß Rekombination allgemein Evolution beschleunigt (Dorit et al.1990, Stemmer 1994, Otto & Barton 1997, Suzuki 1997, Bogarad & Deem 1999). Als eine indirekte Unterstützung dieser Hypothese wird der Befund angesehen, daß viele Proteine dieselben Untereinheiten als Teil ihrer Struktur verwenden. Grundsätzlich können durch nichthomologe Rekombination oder andere Arten von DNA-Transfer (z.B. Transfer durch virale Vektoren) zwei unterschiedliche Proteine bzw. Teile davon miteinander verknüpft werden.
Eine populäre Theorie zur Entstehung neuer Proteine ist Exon shuffling, wobei kleine Proteinabschnitte, welche auf der Genebene von Introns umgeben sind, mit anderen Exons durch intronvermittelte Rekombination neu kombiniert werden (Dorit et al. 1990, Gilbert et al. 1997, Patthy 1999). Es ist experimentell nachgewiesen, daß Verschieben von Polypeptidabschnitten in einigen Fällen gefaltete Proteine erzeugen kann (Riechmann & Winter 2000). Verknüpfung von Polypeptidabschnitten, Transposon-vermitteltes Exon shuffling, Einverleiben von benachbarten Sequenzen, Veränderungen von Leserastern und alternatives Spleißen laufen in der Natur ständig ab (Keese & Gibbs 1992, Long & Langley 1993, Golding et al. 1994, Makalowski et al. 1994, Moran et al. 1999, Finta 2000, Snel et al. 2000, Sorek & Graur 2002). Solche Ereignisse im Genom müssen langsam genug ablaufen, um die Lebensfähigkeit des Organismus nicht zu zerstören. In modernen Organismen scheint die Neukombination von Proteinteilen nicht sehr häufig aufzutreten.
In vielen Untersuchungen wurde das Schicksal von duplizierten Genen verfolgt und beobachtet, wie häufig diese in Populationen fixiert wurden (Ohta 1988, Clark 1994, Hughes 1994, Walsh 1995, Cron et al. 1999, Mazet & Shimeld 2002). Jüngste Ergebnisse zeigen, daß nur eine kleine Zahl der duplizierten Gene nicht in eine Sackgasse führen (Lynch & Conery 2000, Hooper & Berg 2003). Hat ein Gen erst einmal seine Funktion verloren und damit den Schutz durch die Selektion, ist seine Reaktivierung durch Mutationen nahezu unmöglich (Marshall 1994). Es ist auch bekannt, daß viele Gene, die früher Funktionen aufwiesen, diese verloren haben, möglicherweise aufgrund fehlenden Selektionsdrucks (Nishikimi & Yagi 1991, Andersson & Andersson 1999, Mira et al. 2001, Gilad et al. 2003).
Duplizierte Sequenzen haben für Menschen häufig krankhafte Folgen (Mazzarella & Schlessinger 1998). Die hohe Geschwindigkeit, mit der duplizierte Gene ausgelöscht werden, stellt ein Problem dar: es gibt nicht viel Raum für freie Mutationen in redundanten Genen, um neue Funktionen hervorzubringen (Hooper & Berg 2003). Während Genduplikationen möglicherweise zu allmählicher Ansammlung von sinnlosen verdoppelten Genen führen, gibt es wenig Hinweise darauf, daß sich diese zu wirklich neuen Funktionen entwickeln. Verschiebungen und Abweichungen von Funktionen in den Expressionsmustern kommen in der Natur vor (Hughes 1994, Ganfornina & Sánchez 1999, Gu et al. 2002). Genduplikation scheint jedoch kein Weg zur Erzeugung neuer Gensequenzen zu sein. Der Prozeß vergrößert hauptsächlich die Zahl der Varianten in bereits bestehenden Genfamilien. Darüber hinaus bestehen viele Genfamilien nur aus einem einzigen Gen. Im Fadenwurm Caenorhabditis elegans sind 60% der Gene Einzelgene (Semple & Wolfe 1999). Damit kann die Effektivität, mit der durch Genduplikation vollständig neue Gene erzeugt werden, nicht direkt mit derjenigen verglichen werden, mit der Genfamilien erweitert werden.
Viele sequenzierte Genome enthalten eine beträchtliche Anzahl an Pseudogenen und mobilen DNA-Elementen, die grundsätzlich eine Ausgangsbasis für die Herstellung neuer Gene darstellen könnten. Diese DNA-Sequenzen bringen jedoch nicht wirklich neue Gensequenzen hervor, sondern repräsentieren nur einen ziemlich engen Bereich im Sequenzraum moderner Lebensformen. Die Pseudogene beim Menschen, die durch reverse Transkription gebildet werden, entstehen hauptsächlich durch Gene, die stark exprimiert, hoch konserviert und kurz und arm an den Basen G und C sind (Goncalves et al. 2000). Somit ist es unwahrscheinlich, daß diese nichtfunktionellen Genelemente für die Evolution der Vielfalt an Genen in der Natur verantwortlich sind.
Die Geschwindigkeit der Prozesse (Austausch von Aminosäuren, Genduplikation, Exon shuffling), welche möglicherweise neue Proteine hervorbringen, scheinen zu langsam zu sein, als daß sie die Bildung von mehr als zehntausend vollkommen neuen Proteinen in einem unter evolutionären Gesichtspunkten kurzen Zeitraum wie der Kambrischen Explosion und den damit verbundenen Big Bang der Anzahl an Genen erklären könnte..
|
|
Experimentelle Resultate
Durch Evolution nach Darwin kann – per Definition – eine bestimmte, in Lebewesen auftretende Eigenschaft verbessert werden. Die Entstehung einer neuen Funktion ist jedoch ein höchst unwahrscheinliches Ereignis. Moderne Gentechnik hat die Möglichkeit eröffnet, daß nahezu jedes Protein in Zellsystemen produziert werden kann. Einige Proteine werden in Bakterien in großen Mengen produziert, andere in Hefe oder Pilzen und wieder andere in Insekten oder in von Tieren abgeleiteten Zellkulturen. Diese Technologien haben die Möglichkeit zur Herstellung von rein künstlichen Proteinen eröffnet, außerdem erlauben sie die Prüfung von Fragestellungen nach der Häufigkeit von funktionellen Proteinen im Sequenzraum. Ist es möglich, Funktionalität durch einen zufälligen Ansatz zu erreichen? Wenn das möglich ist, dann könnten Darwinsche Mechanismen diese verbessern.
Blanco et al. (1999) untersuchten experimentell den Sequenzraum zwischen zwei kleinen Proteinen mit unterschiedlicher Faltung. Eines besteht aus 62 Aminosäuren und faltet sich als achtsträngiges orthogonales b-Faltblatt „Sandwich-artig“ (vgl. Abb. 3) und das andere Protein besteht aus 57 Aminosäuren und enthält eine zentrale a-Helix, die von einem viersträngigen b-Faltblatt umgeben ist. Die Autoren erzeugten eine Serie von Mutanten und versuchten dadurch zu verstehen, ob es einen evolutionären Pfad von der einen Faltstruktur zur andern geben könnte. Die Schlußfolgerungen ihrer Studie fassen sie folgendermaßen zusammen:
„Der Sequenzraum zwischen SH3 und PG ist enorm groß ... unsere Ergebnisse deuten darauf hin, daß nur ein kleiner Anteil dieses Sequenzraums die entsprechenden Eigenschaften haben würde, um sich in eine bestimmte Struktur zu falten ... Man geht davon aus, daß der erlaubte Sequenzraum für hoch optimierte Sequenzen groß genug sein könnte, um mit anderen Sequenzräumen zu überlappen, die andere Faltungen aufweisen. Dadurch könnte man innerhalb eines denkbaren evolutionären Übergangs eine andere Faltung erreichen. Unsere experimentellen Ergebnisse deuten darauf hin, daß dieses Ereignis sehr unwahrscheinlich ist. Die Natur könnte jedoch andere Mittel haben, um evolvierende Sequenzen, die sich nicht falten, zu bewahren.“
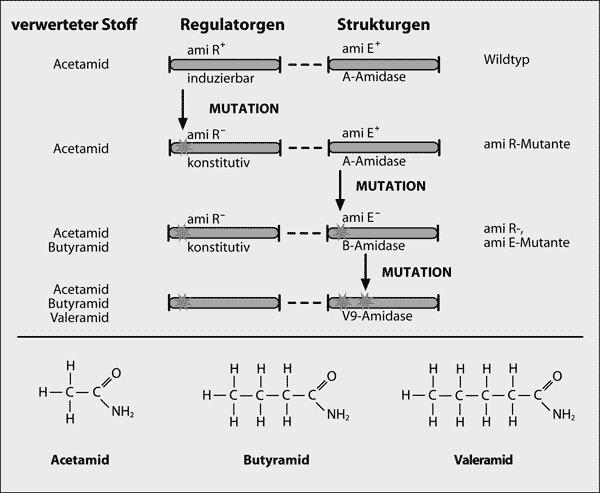 |
Abb. 6: Mutationen zur Erweiterung der Verwertung von Amiden. Durch die Punktmutation ami E– wird beim Bakterium Pseudomonas aeruginosa die A-Amidase (Enzym, das Acetamid spaltet) flexibler, so daß es auch Butyramid spalten kann (B-Amidase). Eine weitere Punktmutation ermöglicht auch noch die Spaltung von Valeramid (V9-Amidase). Die Mutation ami R– bewirkt, daß die A-Amidase nicht nur bei Bedarf (induzierbar), sondern dauernd (konstitutiv) gebildet wird. |
|
Taylor et al. (2001) berichteten, daß aktive Enzyme im zufälligen Protein-Sequenzraum extrem selten sind. Sie benutzten eine Kombination aus intelligenten Entwürfen und evolutionären Mechanismen. Ihre Schlußfolgerung lautet:
„Die geringe Häufigkeit, mit der katalytische Proteine im Sequenzraum auftreten, weisen darauf hin, daß es nicht möglich ist, Enzyme aus unbeeinflußten, zufälligen Bibliotheken in einem einzigen Schritt zu isolieren.“ Keefe & Szostak (2001) entwarfen eine Bibliothek aus 6 x 1012 zufälligen Aminosäuren und selektierten jene, die ATP binden. Sie erhielten schließlich vier neue ATP-bindende Proteine, die keine Ähnlichkeit zu irgendwelchen Einträgen in Proteindatenbanken aufweisen. Sie schlossen ihre Studie folgendermaßen ab:
„Wir vermuten, daß funktionelle Proteine im Sequenzraum ausreichend verbreitet sind (ungefähr 1 pro 1011), so daß sie auf einem völlig stochastischen Weg entdeckt werden könnten, wie es vermutlich war, als Proteine erstmals von Lebewesen gebraucht wurden.“
Wenn diese Schlußfolgerung zuträfe, wären Proteine im Sequenzraum verhältnismäßig verbreitet und können durch Darwinsche Methoden erhalten werden. Allerdings müssen zu dieser Untersuchung einige kritische Anmerkungen gemacht werden. Erstens wurde die „Funktion“ der Proteine nicht zufällig erzeugt und zweitens stellte die „Funktion“ nicht wirklich eine Funktion dar. Es wurde keine katalytische Aktivität gefunden – nur unspezifische Bindung von ATP. Damit erfordert diese „Funktion“ nur ein sehr einfaches Bindungssystem, jedoch keine komplexe dreidimensionale Proteinstruktur. Der letztere Fall (und der ist normalerweise der natürliche) erfordert einen viel größeren Sequenzraum, um ein funktionales Protein zu finden.
Typische Beispiele für Proteinevolution in Bakterien ist die Fähigkeit, neue Substrate zu verwerten. Mutanten von Aerobacter aerogenes entwickelten die Fähigkeit, auf Xylitol zu wachsen (Wu et al. 1968), und Pseudomonas aeruginosa lernte, Buttersäure zu benutzen (Kämpfe 1992; Abb. 6). Sind dies wirklich Beispiele für die Entwicklung neuer Enzymaktivitäten? Die Mechanismen, die zu diesen neu erworbenen Eigenschaften geführt haben, sind keine Beispiele der Art von Evolution, die neuartige Proteine hervorbringt. Im ersten Fall war das Kontrollsystem des Bakteriums durch Mutation inaktiviert worden. Dies ermöglichte eine erhebliche Synthese des Schlüsselenzyms trotz Abwesenheit des natürlichen Induktors. Im zweiten Fall wurde die Substratspezifität eines Amidase-Enzyms leicht verändert, so daß sein aktives Zentrum etwas größer wurde und damit zusätzlich zu Acetamid und Propionamid auch noch Buttersäure hydrolysieren konnte. Es wurde keine neue Faltung oder Aktivität des Proteins erzeugt. Die Flexibilität der Natur erlaubt diese Art von Veränderungen ohne Darwinsche Voraussetzungen.
|
|
Gesteuerte Evolution (directed evolution)
Der Begriff „gesteuerte Evolution“ wird in der Enzymtechnologie für die Erzeugung neuer Proteinvarianten durch zufällige Methoden im Labor verwendet. Powell et al. (2001) beschreibt die Leistungen der Natur folgendermaßen:
„All die herrlichen komplexen Strukturen und Sequenzen, welche die Natur uns bereitstellt, sind durch einen einzigen Prozeß, nämlich den der natürlichen Selektion, entwickelt worden.“
Gesteuerte Evolution beinhaltet eine Anzahl von Techniken, um durch genetische Methoden und Selektion neuer Varianten zufällige Veränderungen an einer Proteinstruktur hervorzubringen. Diese Methoden werden eingesetzt, um modifizierte Enzymkatalysatoren zu erzeugen. Sie sollen den natürlichen Prozeß der Evolution von Proteinen nachahmen. Ist dieser Vergleich zutreffend? Arnold (2003) vom California Institute of Technology beschreibt die notwendigen Bedingungen für den Erfolg einer gesteuerten Evolution folgendermaßen:
1. Die gewünschte Funktion muß physikalisch realisierbar sein. 2. Die Funktion muß auch biologisch und evolutionär ausführbar sein. Praktisch bedeutet das, daß es eine kontinuierliche Reihe von Mutationen vom Ausgangs- bis zum Zielpunkt mit immer besseren Varianten geben muß. 3. Man muß in der Lage sein, Bibliotheken von Mutanten zu erstellen, die komplex genug sind, um seltene nützliche Mutationen zu enthalten. 4. Es muß ein schneller Test oder eine entsprechende Auswahl erfolgen, welche die gewählte Funktion widerspiegelt.
Diese Techniken imitieren zwar Darwinsche Evolution, aber das Problem liegt in der 2. Bedingung: Es muß eine kontinuierliche Reihe von Mutationen mit immer besseren Varianten geben. Das bedeutet, daß die Methode darauf beschränkt ist, bereits vorhandene Funktionen zu verbessern, jedoch nicht dafür geeignet ist, neue Faltungen oder Funktionen hervorzubringen. Arnold erklärt:
„Es ist ein riskantes Abenteuer, eine vollständig neue Funktion entwickeln zu wollen, weil wir kaum wissen, wie weit wir im Sequenzraum gehen müssen, um die neue Funktion zu erzeugen. ... Wenn die neue Funktion den gleichzeitigen Einbau mehrerer Aminosäurereste erfordert, ist es unwahrscheinlich, daß sie in einer zufälligen Bibliothek von Mutanten auftaucht. Ein solches Problem ist vermutlich eher ein Kandidat für eine Kombination aus vernünftigem Design und kombinatorischem Abstimmen.“
Damit „scheint es keine guten evolutionären Wege von einer Faltung zu einer andern zu geben“ (Arnold 1998). Orencia et al. (2001) haben gezeigt, wie Antibiotika-Resistenz durch gesteuerte evolutionäre Techniken hervorgerufen werden kann und wie sie sich vermutlich unter natürlichen Bedingungen bei zunehmendem Stress durch Antibiotika entwickelt. Sie beschreiben die Entwicklung von Antibiotika-Resistenz so:
„Seit der Einführung von Penizillin in den 1940er Jahren haben die Mikroorganismen Resistenz gegen b-Lactam-Antibiotika entwickelt, üblicherweise durch erworbenen oder verstärkten Abbau dieser Medikamente durch die b-Lactamase ... Natürliche Auslese testet nicht alle möglichen Zufallskombinationen von Mutationen, sondern eher durch eine Reihe von Schritten, wobei jeder Schritt zu einem Phänotyp führt, der besser geeignet ist als die Vorgängergeneration ... Natürliche Evolution der b-Lactamasen ereignete sich als Antwort auf eine Reihe verschiedener Penizillinderivate, Cephamycine und vier Generationen von Cephalosporine. Obwohl eine große Anzahl unterschiedlicher Antibiotika in der Klinik verschrieben werden, genügen Mutationen an 13 Stellen (bei einer Gesamtzahl von 290 Aminosäuren) im b-Lactamase Gen, um das breite Spektrum von Antibiotika-Resistenzen hervorzubringen.
Praktische Ergebnisse aus Studien zu gesteuerter Evolution sind beeindruckend, aber begrenzt auf Modifikationen an bestehenden Faltungen und Veränderungen bei vorhandenen Aktivitäten. Es wurden keine neuen Faltungen oder neue Enzymaktivitäten erzeugt.
 |
| Abb. 7: 3-D Strukturen von Xylanase, einem Xylose-Isomerase-Monomer (die dunklen Punkte repräsentieren Metallatome, die für die Aktivität notwendig sind) und ein aktives Xylose-Isomerase-Dimer (v. l. n. r.). |
|
Wir haben in unserem Labor gezielt Design eingesetzt, um einige industriell interessante Enzyme zu verändern. Eines davon ist Xylanase, ein kleines (etwa 200 Aminosäuren) Xylan abbauendes Enzym, das in der Tierfutter-, Papp- und Papierindustrie verwendet wird (Abb. 7). Durch vernünftiges Planen waren wir in der Lage, die Stabilität mindestens 5000mal zu verbessern. Alle bekannten natürlichen Varianten der Xylanasen (11 Familien) haben dieselbe Form, obwohl die Aminosäurezusammensetzung um ca. 60 % variieren kann.
Ein weiteres Enzym, das wir über mehrere Jahre untersucht haben, ist Xylose-Isomerase, das besonders in den USA zur Herstellung von fruktosereichem Maissirup eingesetzt wird. Das Enzym besteht aus vier identischen Untereinheiten. Das aktive Zentrum des Enzyms wird dadurch gebildet, daß sich zwei Untereinheiten miteinander verbinden. Dieses Enzym hat interessante und vorher nicht bekannte Nebenaktivitäten, die zur Herstellung nicht natürlicher Zucker genutzt werden können (Pastinen et al. 1999). Durch geplante Mutation haben wir die Umsatzgeschwindigkeiten einiger schwacher Nebenaktivitäten verstärkt. Allgemein werden solche Nebenaktivitäten durch gesteuerte Evolutionstechniken verbessert, um für die Industrie neue Produktionsmethoden zu entwickeln, die auf Enzymkatalyse basieren.
|
|
Schlu▀folgerungen
Es liegen keine Hinweise vor, daß zufällige chemische Reaktionen geeignet sind, Proteinsekundärstrukturelemente oder ganze Proteine zu erzeugen. Die funktionellen Eigenschaften bereits vorhandener Proteine können durch Planung oder Zufallstechniken umgewandelt oder modifiziert werden. Es ist jedoch bekannt, daß das Design neuer Proteine, für die keine Gegenstücke in der Natur vorliegen, außerordentlich schwierig ist. Dabei sollten sowohl die Proteinstruktur als auch die Funktion geplant werden. Dies unterstreicht ebenfalls die Tatsache, daß funktionelle Proteine im zufälligen Sequenzraum sehr selten sind.
 |
Experimentelle Daten unterstützen die Vorstellung nicht, daß es einen evolutionär wirksamen
Darwinschen Weg von einer Proteinfaltung zu einer anderen gibt. |
 |
|
Entstehung verschiedener Proteinfaltungen und -familien durch Evolution bleibt ein Geheimnis und experimentelle Daten unterstützen die Vorstellung nicht, daß es einen evolutionär wirksamen Darwinschen Weg von einer Proteinfaltung zu einer anderen gibt. Der Protein-Sequenzraum ist enorm, und kein Befund stützt die Annahme, daß funktionelle Proteine in diesem Raum verbreitet sind. Franklin Harold (2001) ergänzt, „wir müssen einräumen, daß gegenwärtig keine detaillierten Darwinschen Darstellungen der Entwicklung von irgendeinem biochemischen oder zellulärem System vorliegen, nur eine Vielfalt sehnsüchtiger Spekulationen“. Dies gilt auch im kleinen Maßstab für den Fall einzelner Proteine. Eine kürzlich erschienene Arbeit faßt die Situation zusammen (Xia et al. 2002): „Die Entwicklung neuer Enzymfunktionen ist von großem Interesse, bleibt aber eine große Herausforderung aufgrund des großen Sequenzraums der Proteine.“
|
Literatur
- Andersson JO & Andersson SGE (1999)
- Insights into the evolutionary process of genome degradation. Curr Opin. Genet. Devel. 9, 664-671.
- Arnold FH (1998)
- When blind is better: Protein design by evolution. Nature Biotech. 16, 617-618.
- Arnold F (2003)
- http://www.che.caltech.edu/groups/fhadirected-evolution.html.
- Blanco FJ, Angrand I & Serrano L (1999)
- Exploring the conformational properties of the sequence space between two proteins with different folds: an experimental study. J. Mol. Biol. 285, 741-753.
- Bogarad LD & Deem WM (1999)
- A hierarchical approach to protein molecular evolution. Proc. Nat. Acad. Sci. USA 96, 2591-2595.
- Bolon DN, Voigt CA & Mayo SL (2002)
- De novo design of biocatalysts. Curr. Opin. Chem. Biol. 6, 125-129.
- Dorit RL, Schoenbach L & Gilbert W (1990)
- How big is the univers of exons? Science 250, 1377-1382.
- Eigen M (1985)
- Makromolecular Evolution: Dynamical ordering in Sequence Space. Ber. Bunsenges. Phys. Chem. 89, 658-667.
- Feistel R & Ebeling W (1982)
- Models of Darwinian processes and evolutionary principles. BioSystems 15, 291-299.
- Finta C & Zaphiropulos PG (2000)
- The human cytochrome P 450 3A locus. Gene evolution by capture downstream exons. Gene 260, 13-23.
- Ganfornina MD & Sánchez D (1999)
- Generation of evolutionary novelty by functional shift. BioEssays 221, 432-439.
- Gilad Y, Man O, Pääbo S & Lancet D (2003)
- Human specific loss of olfactory receptor genes. Proc. Nat. Acad. Sci. USA 100, 3324-3327.
- Gilbert W, de Souza SJ & Long M (1997)
- Origin of genes. Proc. Nat. Acad. Sci. USA 94, 7698-7703.
- Golding GB, Tsao N & Pearlman RE (1994)
- Evidence for intron capture: an unusual path for the evolution of proteins. Proc. Nat. Acad. Sci. USA 91, 7506-7509.
- Goncalves I, Duret L & Mouchiroud D (2000)
- Nature and structure of human genes that generate retropseudegenes. Genome Res. 10, 672-678.
- Gu Z, Nicolae D, Lu HH-S & Li W-H (2002)
- Rapid divergence in expression between duplicate genes inferred from microarray data. Trends Genet. 18, 609-613.
- Harold F (2001)
- The Way of the Cell. Oxford University Press, S. 205.
- Hooper AL & Berg OG (2003)
- On the nature of gene innovation: duplication patterns in microbial genoms. Mol. Biol. Evol. 20, 945-954.
- Hughes AL (1994)
- The evolution of functional novel proteins af Proc. Nat. Acad. Sci. USAter gene duplication. Proc. R. Soc. Lond. B 256, 119-124.
- Hughes MK & Hughes AL (1993)
- Evolution of duplicate genes in a tetraploid animal, Xenopus laevis. Mol. Biol. Evol. 10, 1360-1369.
- Kämpfe L (Hg., 1992)
- Evolution und Stammesgeschichte der Organismen. Stuttgart, S. 98.
- Keefe AD & Szostak JW (2001)
- Functional proteins from a random-sequence library. Nature 410, 715-718.
- Keefe AD & Gibbs A (1992)
- Origins of genes: “Big Bang“ or continouse creation? Proc. Nat. Acad. Sci. USA 89, 9489-9493.
- Lau KF & Dill KA (1990)
- Theory for protein mutability and biogenesis. Proc. Nat. Acad. Sci. USA 87, 638-642.
- Leonov H, Mitchell JSB & Arkin IT (2003)
- Monte Carlo estimation of the number of possible protein folds: effects of sampling bias and folds distribution. Proteins 51, 352-359.
- Long M & Langley CH (1993)
- Natural selection and the origin of jingwei, a chimeric processed functional gene in Drosophila. Science 260, 91-95.
- Lynch M & Conery JS (2000)
- The evolutionary fate and consequences of duplicate genes. Science 290, 1151-1155.
- Makalowski CR, Mitchell GA & Labuda D (1994)
- Alu sequences in the coding regions of mRNA: a source of proteins variability. Trends Genet. 10, 188-193.
- Marshall CR, Raff EC & Raff RA (1994)
- Dollo's law and the death and resurrection of genes Proc. Nat. Acad. Sci. USA 91, 12283-12287.
- Mazet F & Shimeld SM (2002)
- Gene duplication and divergence in the early evolution of vertebrates. Curr. Opin. Genet. Develop. 12, 393-396.
- Mazzarella R & Schlessinger (1998)
- Pathological consequences of sequence duplications in the human genome. Genome Res. 8, 1007-1021.
- McFadden J & Knowles G (1997)
- Escape from evolutionary stasis by transposon-mediated deleterious mutations. J. Theor. Bil. 186, 441-447.
- McLachlan AD (1987)
- Gene duplication and the origin of repetitive protein strucutres. In: Cold Spring Harbor symposium on quantitative biology, vol. LII, Cold Spring Harbor Laboratory, S. 411-420.
- Mira A, Ochman H & Moran NA (2001)
- Deletion bias and the evolution of bacterial genomes. Trends Genet. 17, 589-596.
- Moran JV, DeBerardinis RJ & Kazazian HH Jr. (1999)
- Exon shuffling by L1 retrotransposition. Science 283, 1530-1534.
- Morris SC (2000)
- Evolution: Bringing molecules into the fold. Cell 100, 1-11.
- Nishikimi M & Yagi K (1991)
- Molecular Basis for the deficiency in humans of gulonolactone oxidase, a key enzme for ascorbic acid biosynthesis. Am. J. Clin. Nutr. 54, 1203S-1208S.
- Ohno S (1970)
- Evolution by gene duplication. Springer-Verlag, Heidelberg.
- Orencia MC, Yoon JS, Ness JE, Stemmer WPC & Stevens RC (2001)
- Predicting the emergence of antibiotix resistence by directed evolution and structural analysis. Nature Struct. Biol. 8, 238-242.
- Otto SP & Barton NH (1997)
- The evolution of recombination: removing the limits to natural selection. Genetics 147, 879-906.
- Pastinen O, Visuri K, Schoemaker H & Leisola M (1999)
- Novel reactions of xylose isomerase from Streptomyces rubiginosus. Enzyme Microb. Technol. 25, 695-700.
- Patthy (1999)
- Genome evolution and the evolution of exon shuffling – a review. Gene 238, 103-114.
- Petrov DA, Lozovskaya ER & Hartl D (1996)
- High intrinsic rate of DNA loss in Drosophila. Nature 384, 346-349.
- Powell KA, Ramer SW, del Cardayré SB, Stemmer WPC, Tobin MB, Longchamp PF & Huisman GW (2001)
- Angew. Chem. Int. Ed. 40, 3948-3959.
- Rechenberg I (1973)
- Evolutionsstrategie. Problemata. Stuttgart-Bad Cannstatt.
- Rost B (2002)
- Did evolution leap to create the protein universe? Curr. Opin. Struct. Biol. 12, 409-416.
- Richemann L & Winter G (2000)
- Novel folded protein domains generated by combinatorial shuffling of polypeptide segments. Proc. Nat. Acad. Sci. USA 97, 10068-10073.
- Semple C & Wolfe KH (1999)
- Gene duplication and gene conversion in the Caenorhabditis elegans genome. J. Mol. Evol. 48, 555-564.
- Snel B, Bork P & Huynen M (2000)
- Genome evolution: gene fusion versus gene fission. Trends Genet. 16, 9-11.
- Sorek R, Ast G & Graur D (2002)
- Alu-containig exons are alternatively spliced. Genome Res. 12, 1060-1067.
- Stemmer WPS (1994)
- DNA shuffling by random fragmentation and reassembly: in vitro combination for molecular evolution. Proc. Nat. Acad. Sci. USA 91, 10747-10751.
- Suzuki H (1997)
- The optimum recombination rate that realizes the fastest evolution of a novel functional combination of many genes. Theor. Popul. Biol. 51, 185-200.
- Swetina J & Schuster P (1982)
- A model for polynucleotide replication. Biophys. Chem. 116, 329-345.
- Taylor SV, Walter KU, Kast P & Hilvert D (2001)
- Searching sequence space for protein catalysists. Proc. Nat. Acad. Sci. USA 98, 10596-10601.
- Turunen O, Vuorio M, Fenel F & Leisola M (2002)
- Engineering multiple arginines into Ser/Thr surface of Trichoderma reesi endo-1,4-xylanase increases the temmperature optimum and shifts the pH optimum towards alkaline p.H. Protein Eng. 15, 141-145.
- Xia G, Chen L, Sera T, Fa M, Schultz PG & Romesberg FE (2002)
- Directed evolution of novel polymerase activity: mutation of a DNA polymerase into an efficient RNA polymerase. Proc. Nat. Acad. Sci. USA 99, 6597-6502.
-
|
|
|  |
